8款刘展枝副教授科学研究项目组合作开发应用软件或中文网站如是说
在我看来起微生物重要信息领域的女生物学家,他们肯定会说到一个英文名字,那是刘展枝,一只俊朗的长发也是她的象征。刘展枝副教授合作开发了许多微生物重要信息教育工作者和微生物路径科学人类学家采用的网站和应用软件,是他们嘴中的天神级人物形象,他们此次主要就跟他们如是说一下刘展枝副教授生物医学合作开发过的中文网站和应用软件。
大背景如是说
他们先简单了解下刘展枝副教授,1992 年考进清华大学微化学系,2002 年于麻省理工学院取得微生物科学重要分子生物教授和软件工程双学位教授学位后, 被直接任为耶鲁大学大学有名无实现职副教授,目前同时供职于美国耶鲁大学大学及Dana-Farber肺癌科学研究院,Fig 1是刘展枝副教授的网页,邮箱是https://www.dfhcc.harvard.edu/insider/member-detail/member/xiaole-shirley-liu-phd/,2018年成为AACR历史上第一位在主全会上刊登演说的华裔男性科学家,见Fig 2。
刘展枝副教授的科学研究浓厚兴趣主要就包括五个大的方面,Cancer Epigenetics,CRISPR screens,Cancer Immunology,Single Cell,并且也刊登了许多高质量的该文,Fig 4列出了刘展枝副教授近些年来的一些该文。
Fig 5是他们今天的主人公,这那哥也是刘展枝副教授在最近的微生物重要信息合作开发人员讨论会上讲自己最捷伊COP1这个DNA工作的情况Seiches出来的,当日全会的氛围感觉很酷,刘展枝副教授一直在致力新领域、新科学研究路径的合作开发,此次做的是通过一种捷伊方法来批量研制潜在性抗生素。上面他们将按照次序逐个如是说下刘展枝副教授合作开发的这些新浪网中文网站或者应用软件的采用。
中文网站和应用软件
TRUST4
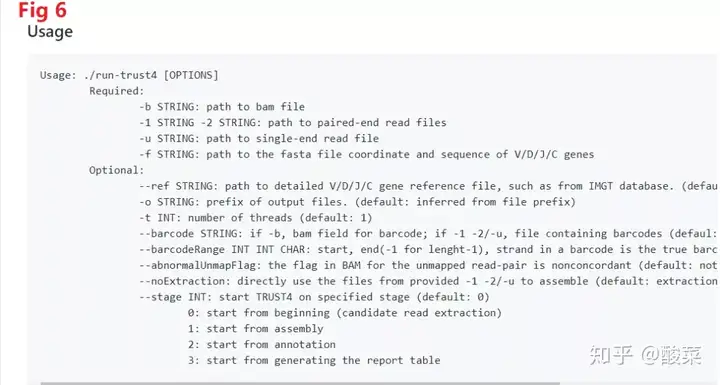
第一个工具是TRUST4,邮箱是https://github.com/liulab-dfci/TRUST4,这个工具他们应该显得稍微陌生点,这个工具主要就是用来分析实体组织,包括肿瘤组织的RNA测序的数据的TCR和BCR序列,这部分内容其实也比较火热,主要就是免疫组库(Immune Repertoire,IR)的内容,免疫组库指的是机体内 T 淋巴细胞和 B 淋巴细胞多样性的总和,反映机体免疫系统在特定时间段内应对外界刺激应答的能力。可以通过检测免疫组库来检测患者的免疫能力的高低,TRUST4主要就是通过对高可变的V, J, CDNA的可变区域进行de novo assembly,并通过比对免疫资料库IMGT的序列,得到样本的TCGR和BCR序列的多样性,TRUST4可以接受单端或者双端的测序数据,并且不限制测序的read长度。他们可以直接通过git克隆代码库就可以采用这个应用软件了,应用软件参数见Fig 6
TIMER
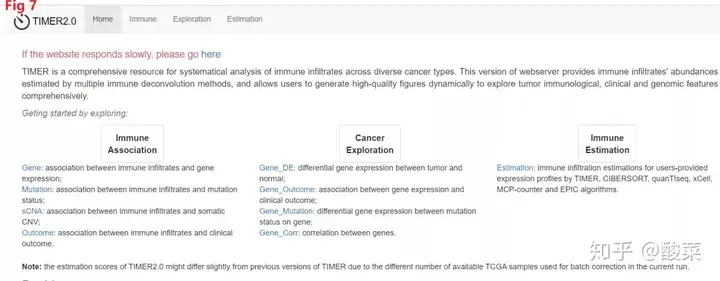
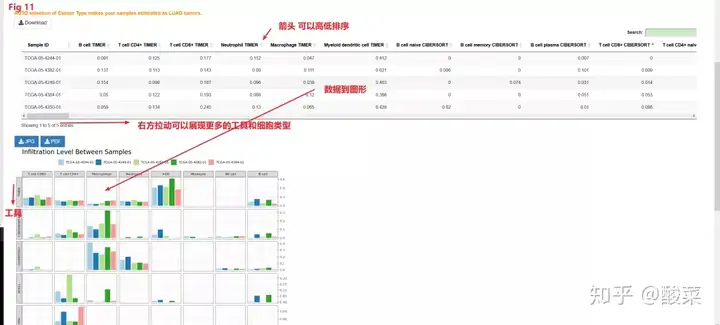
第二个是一个在肿瘤,免疫领域被他们广泛引用的中文网站TIMER,这个中文网站分为2个版本,https://cistrome.shinyapps.io/timer/TIMER: Tumor IMmune Estimation Resource;http://timer.cistrome.org/TIMER2.0。TIMER于2017年刊登在Cancer Research上,截止至2020.08.14已经被引用431次;TIMER2.0于2020年刊登在Nucleic Acids Research,截止至2020.08.14已经被引用5次。TIMER2.0相对于TIMER来说最大的改变是,TIMER只采用了一个单一的算法,但是在TIMER2.0采用了更多的更稳健的算法来评估TCGA资料库或者采用者自己提交的表达数据,也是结果更准确,更具有更可靠的微生物学意义,而且TIMER这个版本1网页长时间不点击就会自动断开。如Fig 7所示。
TIMER2.0主要就包括3个模块,Immune Association,Cancer Exploration,Immune Estimation。其中Immune Association主要就包括四大子模块,Gene,Mutaion,sCNA,Outcome;Cancer Exploration主要就包括四大子模块Gene_DE,Gene_Outcome,Gene_Mutation,Gene_Corr;Immune Estimation只包括一个子模块Estimation。
上面他们每个大模块抽取一个示范一下采用。他们进入每个模块点击页面最上方的这一排的图标进入。
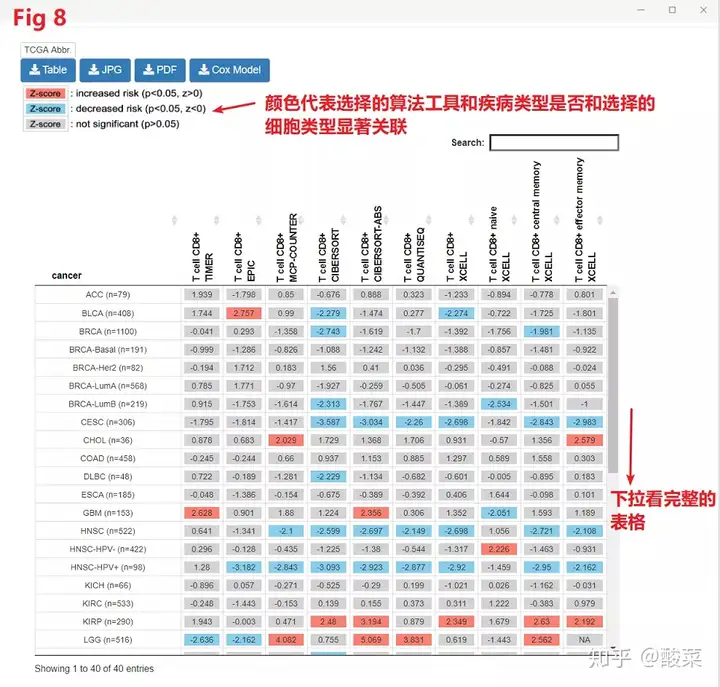
在Immune Association他们选择子模块Outcome。他们就选择默认参数,点击submit参数即可返回得到结果。根据图中的标注,他们可以看到采用EPIC这个工具,在BLCA这个肿瘤中,T cell CD8+是一个显著的增加风险的细胞类型。
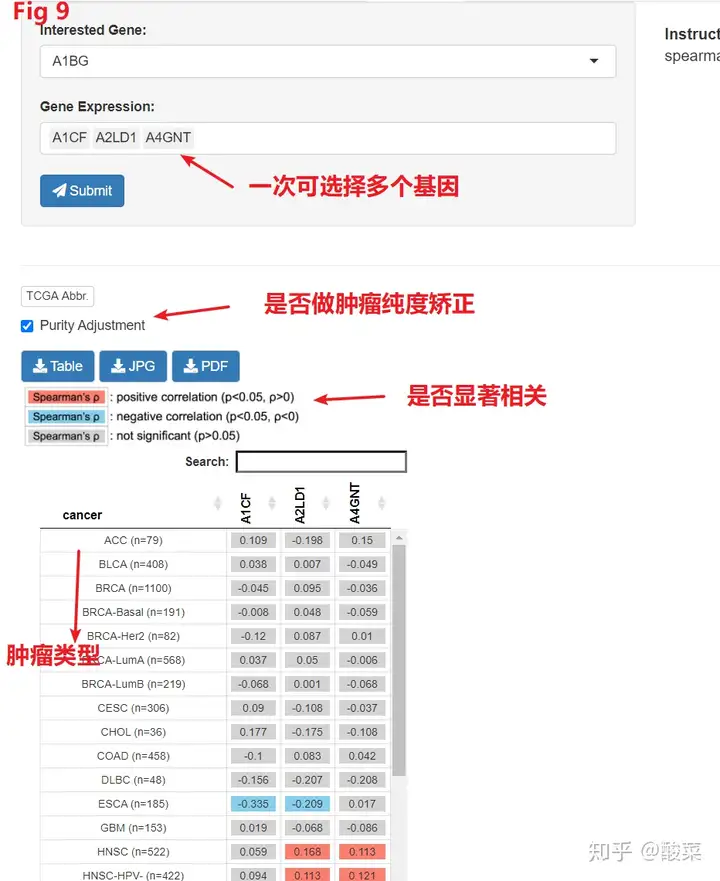
在Exploration模块他们选择Gene_Corr这个子模块,在Gene Expression这个选项,他们可以一次选择多个DNA去和他们的Interested Gene做相关性计算,并且他们可以决定是否勾选Purity Adjustment,这个主要就是根据他们是否得到了自己所需要的结果进行调整。
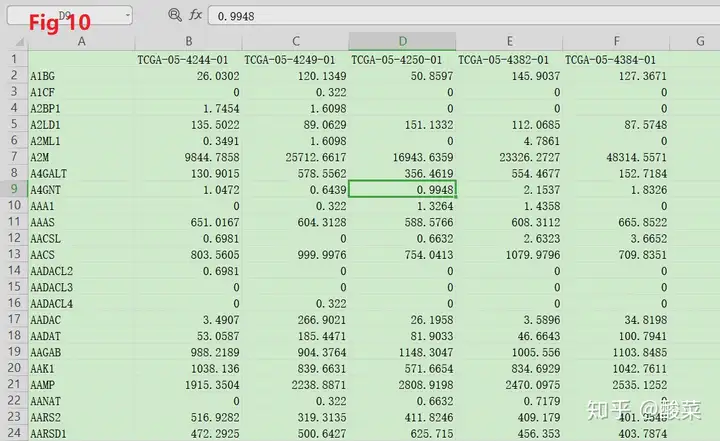
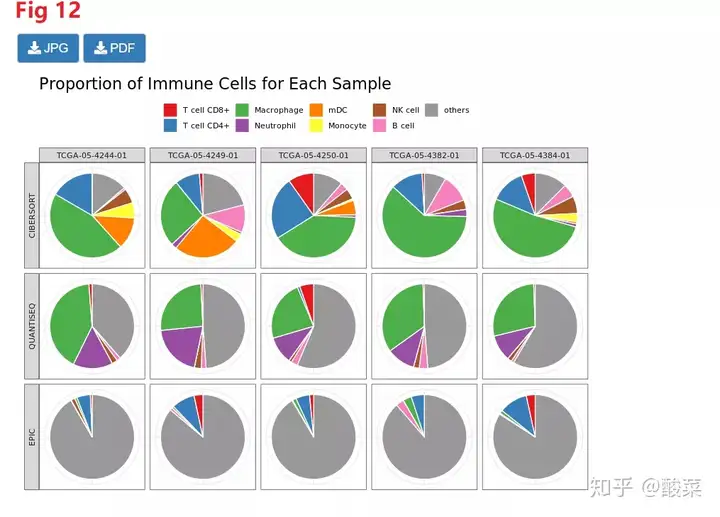
在Esimation模块,他们可,就会在右侧下方看到进度条,google浏览器没成功的话可以选择火狐浏览器。Fig 10是他们的示例数据,一共5个样本。得到的结果一共分为2个图片讲解。Fig 10他们可以看到检测到数据是LUAD类型,跟他们下载的是一致的肿瘤类型。Fig 12是每个细胞在预测工具得到的在每个样本的比例。这些结果可以跟着这些细胞代表的特殊的微生物学意义来说明他们科学研究的内容的一些特殊的属性。
TIDE
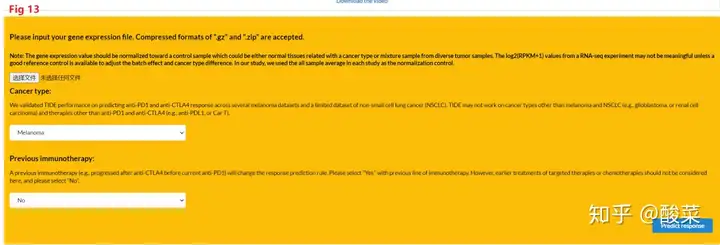
TIDE资料库的全称是Tumor Immune Dysfunction and Exclusion,翻译为中文是肿瘤免疫功能障碍和排斥,中文网站地址是http://tide.dfci.harvard.edu/,而且这个中文网站必须注册才能采用。这个资料库他们平时用到的可能性比较小,因为一般人也拿不到这样的数据,里面主要就用到的数据集是TCGA和PRECOG。他们知道目前通过免疫检查点抑制剂(immune checkpoint blockade,ICB)的肿瘤治疗只有一部分人可以长期获益,这个中文网站是用来推测调节肿瘤免疫的DNA的功能,用来综合分析免疫功能障碍和排斥对两种肿瘤(黑色素瘤和非小细胞肺癌)的免疫逃逸机制,从而来有效预测免疫检查点抑制治疗的效果。所以这个资料库针对的肿瘤是有限的。中文网站的截图如Fig 13。
TISCH
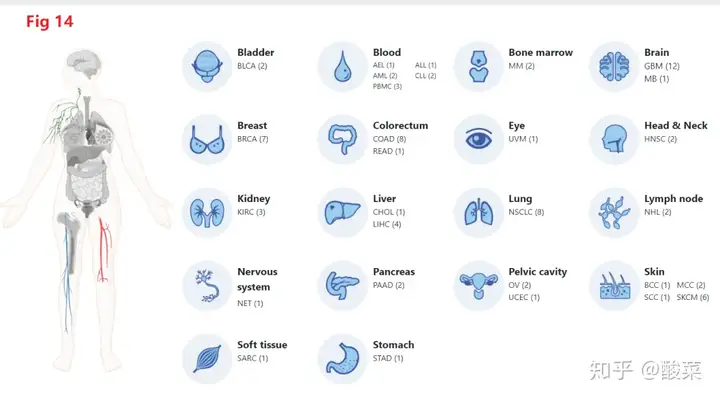
http://tisch.comp-genomics.org/home/ 。一共包括79个数据集,大约200万个细胞。Fig 14描述了这个中文网站的数据集的各个肿瘤的组成,比如BLCA有2个数据集。
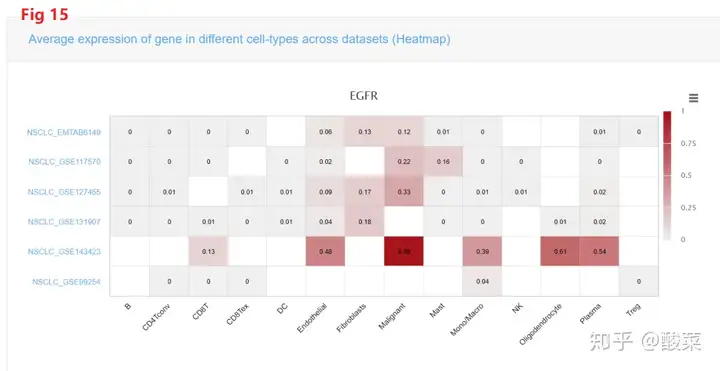
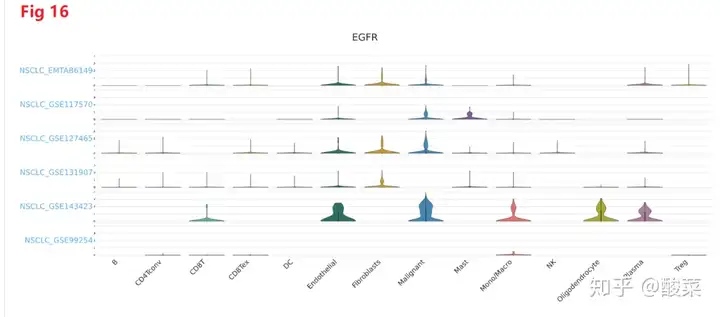
他们还可以搜索单个关心的DNA在不同数据集的的不同细胞类型的平均表达,作者在这里也不建议选择所有的肿瘤类型,因为那样太慢了。并且数据集的个数也可以自己勾选。Fig 15是热图,颜色越深,说明表达量越高,Fig 16是小提琴图,根据小提琴图的高度也可以反映表达量的高低。
Cistrome Data Browser
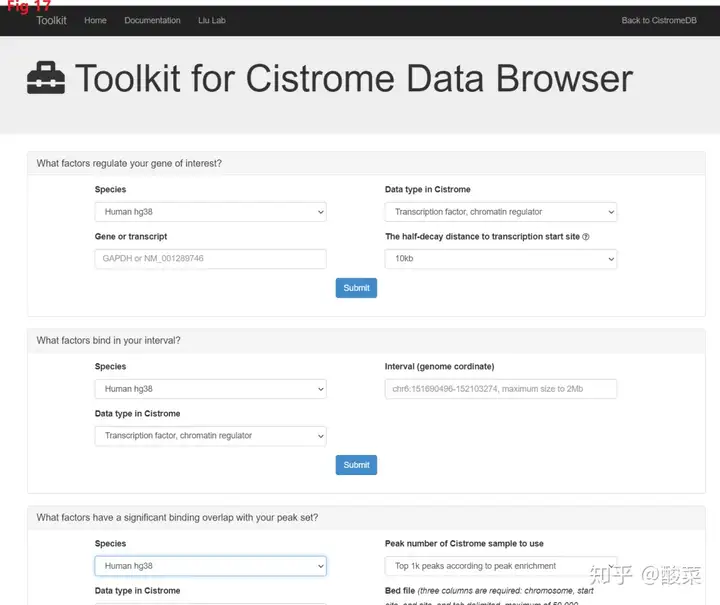
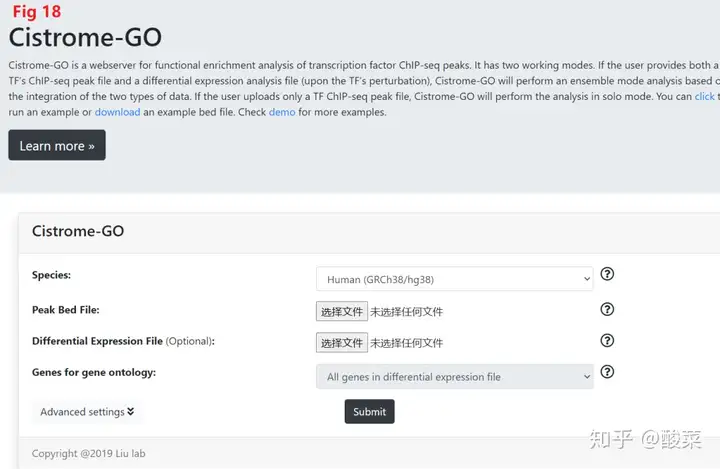
Cistrome Data Browser是一个科学研究顺势调控元件的资料库,中文网站地址是http://cistrome.org/db/#/域被什么转录因子调控,就可以采用CistromeDB Toolkit,中文网站地址是http://dbtoolkit.cistrome.org/,但是这个没法选择不同的参考DNA组版本,如图Fig 17。如果你有转录因子的CHIP-seq的数据,就可以利用Cistrome-GO来科学研究这个转录因子潜在性的位置功能,中文网站地址是http://go.cistrome.org/,并且可以选择不同的参考DNA组版本,如图Fig 18。
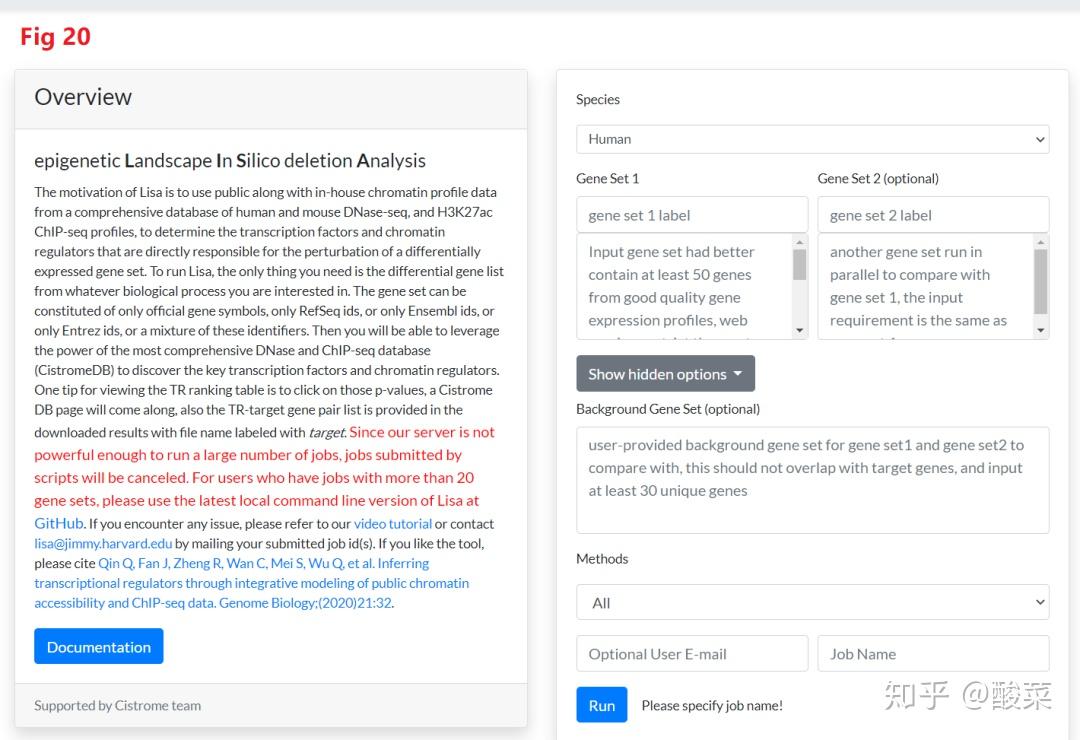
LISA
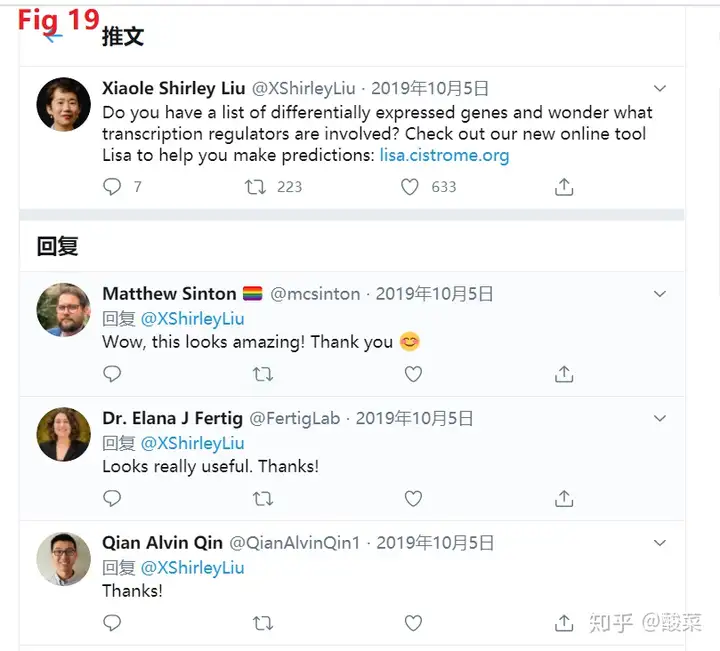
LISA主要就是可以用来当你有差异表达的DNA,并且希望了解有什么转录因子可能参与了这个过程,那么你就可以采用这个工具,这也是刘展枝副教授在推特的原话,如图Fig 19
如图Fig 20这个中文网站可以用来做人和小鼠的数据科学研究,至少需要属于一个DNA组,不少于50个结果会更加准确,也可以自己输入大背景DNA集,还可以选择不同的方法,并且可以设置邮箱来提醒结果。
MAESTRO
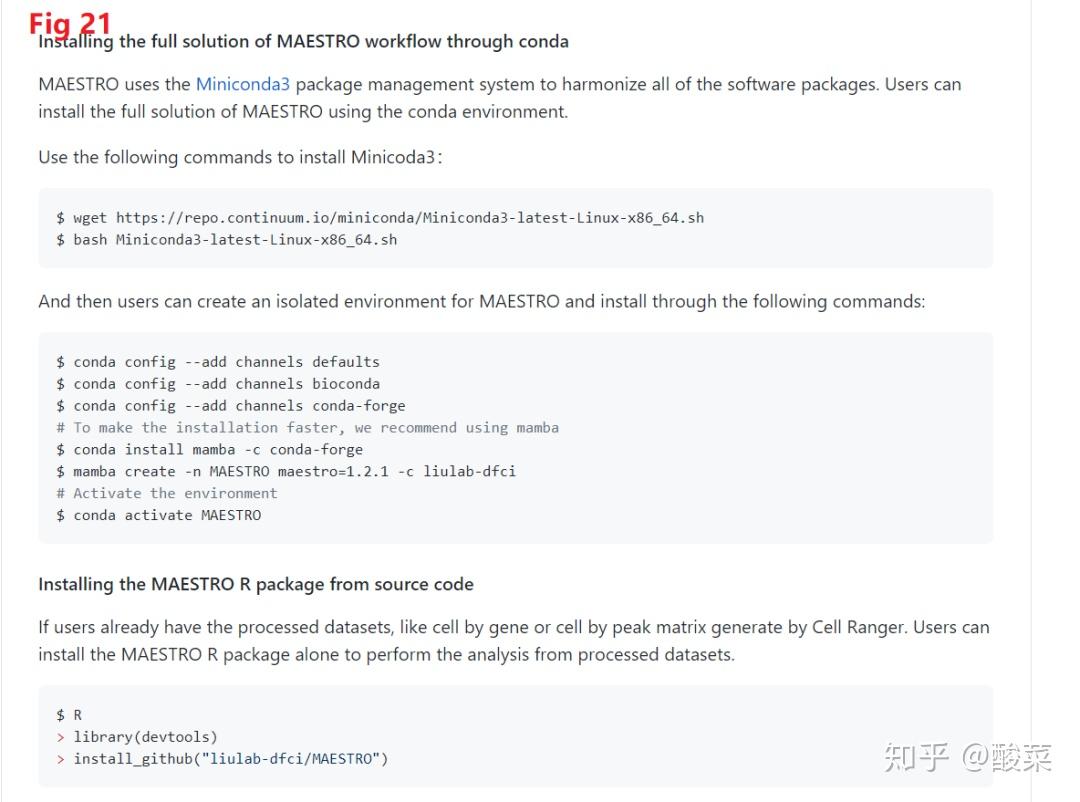
MAESTRO是一个应用软件,全称是Model-based AnalysEs of Single-cell Transcriptome and RegulOme,可以在https://github.com/liulab-dfci/MAESTRO看到,是一个分析单细胞转录组数据和ATAC-seq数据的流程,采用snakemake编写,可以从fastq数据开始处理,可以做数据质控,差异分析,聚类,调控网络分析,目前支持Smart-seq2, 10x-genomics, Drop-seq, SPLiT-seq 的转录组数据; microfudics-based, 10x-genomics , sci-ATAC-seq的scATAC-seq数据,可以通过conda便捷的安装,并且版本更迭一直在做,应用软件只能在linux运行,需要Python (>= 3.0)和R (>= 3.6.1)支持,安装过程见Fig 21
MAGeCKFlute
MAGeCKFlute是一个用来分析批量CRISPR敲除DNA功能科学研究的一个R包,存放在bioconductor。这个可能一般人也用得上,这个实验做起来也不便宜。其实之前刘展枝副教授已经合作开发了MAGeCK and MAGeCK-VISPR来科学研究CRISPR的相关科学研究。更细致的描述可以参考Fig 22。
CARE
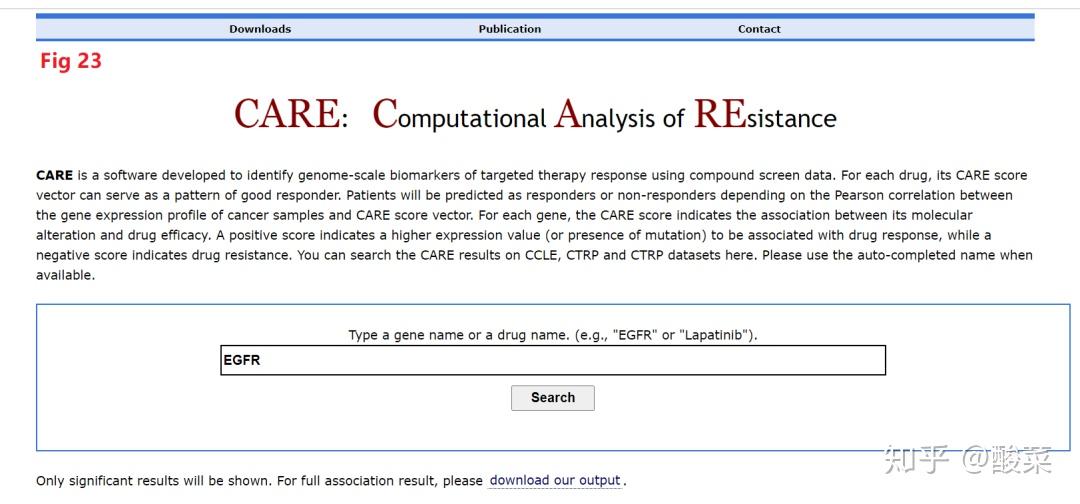
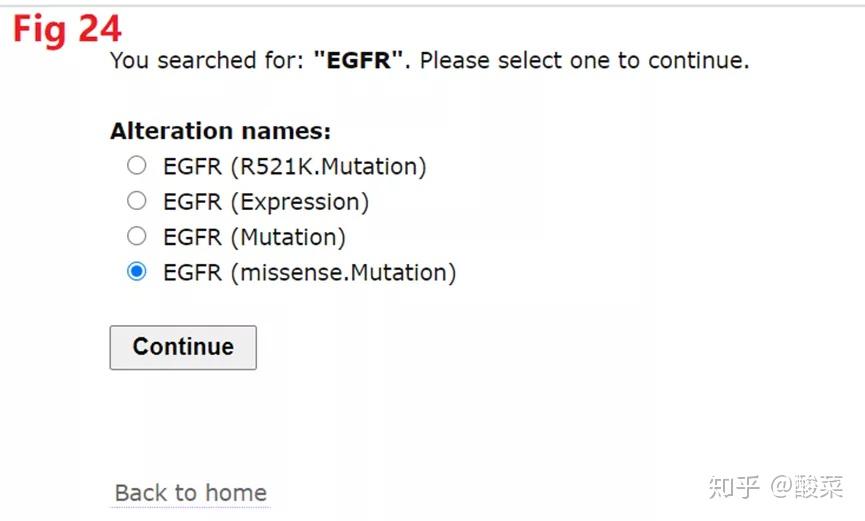
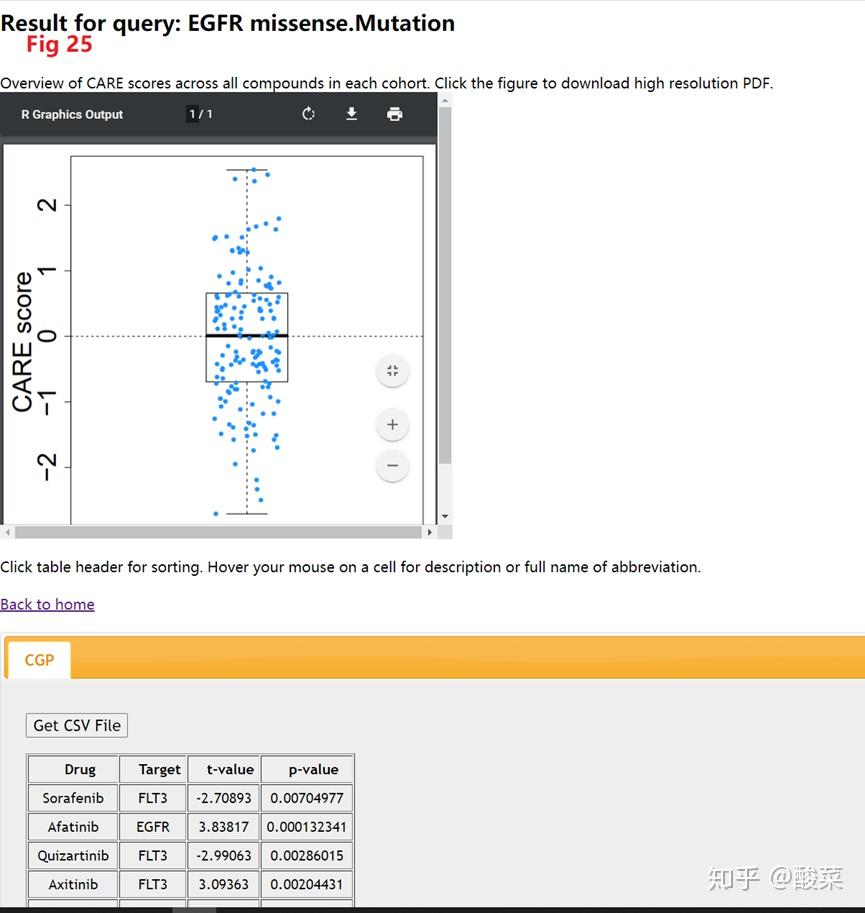
CARE主要就是来预测靶向治疗和耐药的调节DNA,全称是Computational Analysis of Resistance,邮箱是http://care.dfci.harvard.edu/,如图在Fig 23他们输入EGFR之后,会跳出如图Fig 24要求他们进一步筛选,比如我们选择最后一个,然后就可以得到中文网站的评分结果,如图Fig 25,图片可以通过PDF下载,表格数据可以下载CSV格式,这个就需要他们有一些临床用药的知识,不一定每个人都能看懂这个意思。
在本篇该文中,他们有详细,也有简略的如是说了刘展枝副教授合作开发的一些应用软件和中文网站,主要就是围绕肿瘤,但是他们也可以看到刘展枝副教授科学研究的领域幅度之广阔,令人真是佩服。
转载请注明:解螺旋·临床医生科研成长平台
点击上面的链接可扫码添加我的现职微信,私信不回问题:
传播科研最前沿知识;
中国临床医生科研成长平台;
现在添加还可领取10G科研资源大礼包!(内含:SCI写作投稿技巧、资料库采用讲义、国自然工具包……)